Forschungsgebiete
Der Forschungsbereich von Prof. Dr. Dieter Jendrossek beschäftigt sich mit dem Metabolismus von Biopolymeren in Mikroorganismen. In der Arbeitsgruppe werden drei Themenkomplexe bearbeitet:
Metabolismus von Polyhydroxybuttersäure (PHB) in Bakterien: Nach der Erarbeitung der biochemischen Grundlagen des extrazellularen Abbaus und der intrazellulären Wiederverwertung des Speicherstoffes PHB durch PHB Depolymerasen (PhaZs) besteht der derzeitige Schwerpunkt in der Erforschung der Biogenese von PHB Granula, sog. Carbonosomen, in dem Knallgasbakterium Ralstonia eutropha H16 sowie in der Produktion von PHB als Biokunststoff durch Kultivierung von Bakterien auf Abfallströmen der grünen Papierindustrie in Kooperation dem IBVT und einem lokalen Hersteller von Zellulosefasern.
Abbau und enzymatische Spaltung von Kautschuk (Poly-cis-1,4-isopren): In einem zweiten Forschungsschwerpunkt wird der Abbau und die enzymatische Spaltung von Kautschuk (Poly-cis-1,4-isopren) durch bakterielle Dihäm-Dioxygenasen (Rubber Oxygenasen RoxAs, RoxBs und RoxCs) in gramnegativen kautschukabbauenden Bakterien bzw. durch das Latex Clearing Protein (Lcp) in grampositiven kautschukabbauenden Bakterien untersucht.
Subzelluläre Struktur und Lokalisation von Polyphosphaten: Der dritte Forschungsbereich untersucht die subzelluläre Struktur und Lokalisation von Polyphosphaten, den sogenannten Polyphosphatosomen, in Abhängigkeit vom Zellzyklus sowie die Funktionen der mit den Polyphosphatosomen assoziierten Proteine in Ralstonia eutropha und Agrobacterium tumefaciens mit Hilfe molekularbiologischer und life cell imaging Techniken.
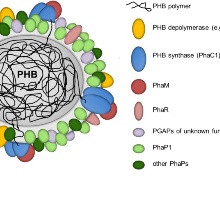
Polyhydroxybuttersäure (PHB) und verwandte Polyhydroxyalkanoate (PHAs) werden von vielen Bakterien als Speicherstoffe in Form von PHA-Granula synthetisiert. PHAs sind sogenannte „Biokunststoffe“. Sie gelten aufgrund ihrer biologischen Abbaubarkeit zu H2O und CO2 und ihrer Synthese aus nachwachsenden Rohstoffen als umweltfreundliche Alternative zu herkömmlichen Kunststoffen auf Rohölbasis. PHA-Granula mit ihrer komplexen Oberflächenstruktur aus verschiedenen Proteinen stellen kleine funktionelle Einheiten (Organellen) dar, für die wir den Begriff „Carbonosomen“ geprägt haben. In unserer Arbeitsgruppe untersuchen wir die an der subzellulären Bildung von PHB/PHA Granula beteiligten Proteine und ihre spezifischen Funktionen. Dabei kommen insbesondere molekularbiologische Methoden und Life-Imaging-Techniken zum Einsatz.
- Aufbau von Bioplastik aus Abfällen der Papierherstellung
Beim Themenbereich Polyisopren (Kautschuk, Gummi) steht die Frage im Vordergrund, wie Kautschuk (Autoreifen!) durch mikrobielle Enzyme angegriffen und schließlich abgebaut (mineralisiert) wird. In den letzten Jahren haben wir eine neuartige Häm-haltige Dioxygenase (Rubberoxygenase RoxA) entdeckt und kürzlich deren Struktur aufgeklärt. Derzeit untersuchen wir den oxidativen Reaktionsmechanismus mit biochemischen, biophysikalischen und molekularbiologischen Methoden.
- Metabolismus von Citronellol und anderen methylverzweigten Verbindungen
- das Thema ist seit 2014 abgeschlossen
Polyphosphat (PolyP) ist ein anorganisches Biopolymer, das in allen Organismen vorkommt und daher von fundamentaler Bedeutung ist. PolyP-Granula sind komplexe Gebilde (Polyphosphatosomen), die aus einem PolyP-Kern und daran gebundenen Proteinen (z.B. PolyP-Kinasen, Phosinen etc.) bestehen. Wir untersuchen die Struktur, die Bildung und die spezifischen Funktionen von PolyP Granula mit molekularbiologischen Methoden und Life Imaging Techniken in Agrobacterium tumefaciens und Ralstonia eutropha als Modellorganismen.
Kontakt